Le plan d’action 2018-2021 de l’IFB inclut un volet innovant axé sur la bioinformatique intégrative (WP2), visant à assurer la cohérence et la pertinence des développements et services en bioinformatique, ainsi que leur lien avec les initiatives européennes (ELIXIR, appel inter-ESFRI). Dans ce contexte, le défi majeur, avec ces projets pilotes, est de stimuler le développement d’outils et de services permettant d’intégrer des données hétérogènes à haut débit (séquençage, imagerie, protéomique, métabolomique, etc.) afin d’abord la complexité des systèmes biologiques. La pertinence de ces approches doit être démontrée en les appliquant à des cas d'utilisation spécifiques reposant sur des données produites par au moins deux infrastructures de recherche nationales, des installations de modèles animaux, des cohortes humaines ou des plans nationaux.
Les projets doivent également correspondre aux priorités nationales, notamment la santé, la sécurité et la sûreté alimentaires, l'environnement, la biotechnologie et la recherche fondamentale. Les projets pilotes ont été conçus pour obtenir des résultats a relativement court terme en s’appuyant sur le know-how existant de différentes structures du PIA (infrastructures, cohortes, plans). L’appel à lettre d’intention était formulé de la façon suivante: dans une période de 18 à 24 mois, répondre à une question de recherche spécifique mettant en oeuvre une approche de bioinformatique intégrative illustrée par un cas d’étude précis impliquant plusieurs infrastructures nationales. Les candidatures retenues ont été soutenues par un contrat à durée déterminée de deux ans maximum.
Les projets pilotes impliquent dix infrastructures de recherche nationales (IFB, France Génomique, MetaboHub, ProFI, FRISBI, FBI, FLI, Phenome, EMBRC-Fr, F-CRIN), deux installations de modèles pour les animaux et des cohortes humaines (PHENOMIN, BIOBANQUES) et le plan national “France Médecine Génomique 2025”.
Cinq projets ont été sélectionnés:
Les approches de la protéomique et de la métabolomique fournissent des informations uniques et complémentaires pour déchiffrer la fonction des gènes, élucider les phénotypes et découvrir avec robustesse de nouveaux biomarqueurs pour le traitement des maladies. Des ressources bioinformatiques nationales ont été récemment développées par les infrastructures IFB, ProFI et MetaboHUB pour analyser chacune de ces données en omique. Nous proposons ici de développer de nouveaux outils permettant l'analyse de données protéomiques et métabolomiques à haut débit et combinées. Tout d'abord, la modélisation statistique sera utilisée pour explorer les informations spécifiques et communes des différentes données, et pour déterminer comment leur combinaison en tant que signatures moléculaires peut interpréter et prédire les phénotypes de manière optimale. Deuxièmement, l'intégration des réseaux sera utilisée pour faciliter l'interprétation et l'annotation des données. Les approches et outils mathématiques seront validés sur deux cas d'utilisation portant respectivement sur le phénotypage à haut débit de modèles de souris générés par l'infrastructure PHENOMIN, et la découverte de nouvelles fonctions des gènes dans les approches de microbiologie systémique développées par France Génomique. Le projet fournira à la ressource bioinformatique nationale Workflow4Metabolomics des outils et des flux de travail innovants pour l'analyse combinée à haut débit des données protéomiques et métabolomiques, et ouvrira la voie à de nouveaux services de phénotypage fournis aux universités et à l'industrie. Il enrichira également la base de données PHENOMIN IMPC de modèles de souris et la base de connaissances microbiennes MicroScope.
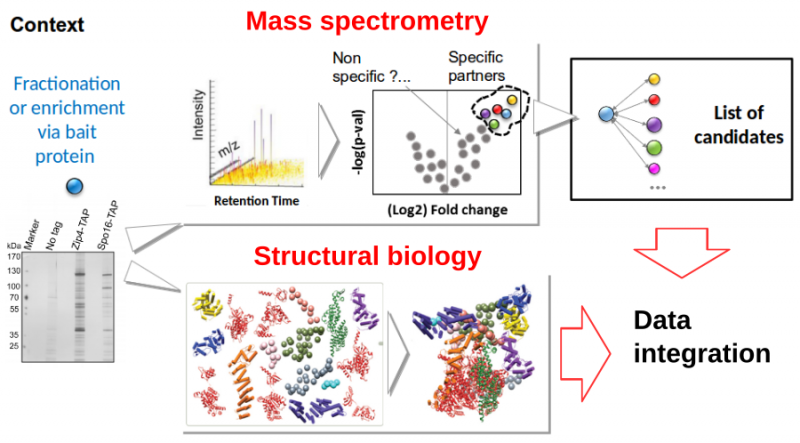
La spectrométrie de masse est désormais une technique essentielle pour la caractérisation des espèces moléculaires et de leurs interactions. Elle a récemment acquis une extrême précision ainsi qu'un important débit tout en ne requérant que peu de matériel. Pour de nombreuses questions biologiques, les analyses de protéomique ou d'interactomique constituent désormais des points d’entrée incontournables. Toutefois, ces études s’arrêtent souvent à l’étape d’énumération des macromolécules en interaction (protéines principalement) sans pousser plus avant l'analyse des séquences identifiées. Or, les aspects structuraux ou évolutifs offrent une grille d’interprétation puissante pour les biologistes (interprétation de mutations chez les patients perturbant les assemblages, mutagenèse ciblée et dissection fonctionnelle, criblage virtuel, etc…). Nous proposons de réunir et combiner des outils de bioinformatique haut débit pour la modélisation des structures de protéines et de leurs assemblages. Le service déployé permettra aux utilisateurs de PROFI de faciliter le post-traitement des cribles et d’amplifier l’information exploitable. Il bénéficiera en même temps aux utilisateurs de FRISBI pour la modélisation intégrative des données acquises sur leurs plateformes. Le couplage entre ces deux grandes stratégies de caractérisation des interactomes sera potentialisé par l’environnement bioinformatique offert par l’IFB. Il bénéficiera à l’ensemble de la communauté des biologistes travaillant sur les interactions entre macromolécules améliorant, par exemple, l’analyse des dysfonctionnements pathologiques (modification des interactions, isoformes, etc…).
Phenome ou Emphasis-fr, l’infrastructure nationale de phénotypage végétale, dispose de neuf nœuds, ou plateformes expérimentales, répartis sur le territoire national. Chacun de ces nœuds dispose d’un système d’information dont en particulier PHIS, actuellement en production sur deux nœuds et en cours de déploiement sur d’autres. Un de ces nœuds, HiTMe, prend en charge le phénotypage métabolique d’échantillons prélevés sur les autres nœuds. C’est également l’une des 4 composantes de la plateforme Métabolome Bordeaux, elle-même composante de l’infrastructure MetaboHub. L’intégration des données produites par ces différents nœuds repose sur les standards construits conjointement par Emphasis et Elixir. Elle implique en particulier la définition d’ontologies de variables phénotypiques et environnementales, la définition du standard MIAPPE et son implémentation via les web services de la Breeding API implémentés dans PHIS et dans GnpIS, Système d’Information INRA appartenant à Elixir-fr permettant la publication de données de phénotypage végétal. L’objectif du présent projet sera de permettre la consolidation et l’intégration de différents jeux de données Maïs, obtenus à travers plusieurs projets scientifiques, afin de permettre l’exploration de l’impact réciproque des conditions environnementales (pratiques culturales, conditions pédoclimatiques, …) et du choix des variétés de maïs sur la synthèse de différents métabolites d’intérêt.
Datasets publiés
Publication en rapport avec le projet
Autre contribution
Trois projets supplémentaires ont été réorientés pour être soutenus par d’autres actions de l’IFB: “Soutien aux bases de données”, “Catalyser l’interopérabilité” et “Partage de services avec les infrastructures de recherche nationales”:
Porteur : Faisal Bekkouche (EMBRC, Villefranche/mer)
Structures du PIA : EMBRC, FBI
EMBRC-Fr/EU est une infrastructure d’excellence fournissant l’accès aux ressources biologiques marines, leurs écosystèmes, et aux techniques d’analyse de ces ressources marines pour tout utilisateur public/privé du monde entier. Le nœud français d’EMBRC-ERIC propose des stratégies d’imagerie d’organismes marins de haute technologie (imagerie live, imagerie 3D et imagerie à haut débit) sur les trois sites de l’infrastructure (Institut de la Mer de Villefranche, Observatoire Océanologique de Banyuls et Station Biologique de Roscoff). Les progrès récents dans le domaine de l’imagerie ont fait croître de manière exponentielle la quantité d’informations recueillies par les centres producteurs de données. L’exploitation optimale de ces données est essentielle pour débloquer l’application des modèles marins à des nombreux champs de recherche et d’application, ainsi que pour l’exploitation et la préservation des écosystèmes et de la biodiversité marine. La mise en place du webservice My-EMBRC-IMAGE associé à une infrastructure de stockage distribuée accessible à distance, permet alors la sauvegarde, la gestion, la visualisation et l’analyse d’images de microscopie. Ce projet s’appuyant sur une synergie entre les infrastructures EMBRC-France, FBI et IFB doit accroître leur rayonnement et leur impact auprès des communautés scientifiques et du public.
Porteur : Pascal Roy (IFB PRABI, Lyon)
Structures du PIA : France Génomique, F-CRIN
Le projet s’inscrit dans le programme 2018-2021 de l’IFB, répondant plus précisément à l’objectif d’intégration des ressources et de renforcement de l'interopérabilité entre équipes. Ce projet structurant consiste en la mise en place d’un réseau d’équipes expertes pour l’analyse statistique des données de grande dimension et l’interprétation contextualisée des résultats de l’analyse du séquençage parallèle massif du génome humain et des agents infectieux. Le projet contribuera à l’implémentation des méthodes d’analyse statistiques développées par les équipes partenaires sur les différentes plateformes d’une part, au développement et à la validation de nouvelles méthodes d’analyse statistique dans le cadre de collaborations entre ces équipes d’autre part. Le projet porte sur l’analyse statistique des performances des pipelines de séquençages, l’étude des propriétés des modèles d’identification de biomarqueurs diagnostiques, pronostiques et théranostiques, et le développement de modèles intégrant les composantes biologiques et expérimentales de la variabilité. L’accent est mis sur l’inférence statistique et l’étude des propriétés prédictives des modèles dans le cadre de leurs applications en épidémiologie et en médecine clinique. L’action structurante associe la diffusion des méthodes, et la mise en place de projets collaboratifs entre les plateformes.
Porteur : Christophe Beroud & David Salgado (Inserm, Marseille)
Structures du PIA : -
Dans le cadre du réseau national de diagnostic des maladies génétiques, du Plan France Médecine Génomique 2025 et de la communauté Human Data d’ELIXIR, la collecte et l’annotation standardisée des Copy Number Variation (CNV) constitutionnelles retrouvées chez les patients présente un intérêt majeur pour faciliter l’identification de nouveaux gènes responsables de maladies génétiques (recherche), l’identification de CNV pathogènes et faciliter l’interprétation médical avec diminution du nombre de variant de signification inconnue (Variant of Unknown Significance- VUS) (diagnostic). La France via la plateforme GMGF-GBIT de Marseille, est leader de la communauté “Human CNV” d’ELIXIR et développe la base de données nationale BANCCO en partenariat avec le réseau national des laboratoires de diagnostic AchroPuce qui regroupe l’ensemble des laboratoires réalisant des Analyses Chromosomiques sur Puce à ADN (ACPA ou CGH array) dans un cadre diagnostic pour des pathologies constitutionnelles. Ce développement est réalisé avec l’équipe des Hospices Civils de Lyon dirigée par le Professeur Sanlaville qui est également co-responsable de la plateforme AURAGEN du Plan France Médecine Génomique 2025. Le professeur Christophe Béroud (GMGF-GBIT) est également impliqué dans le Plan France Médecine Génomique 2025 où il est l’un des experts du comité CAD (Collecteur et Analyseur de Données). A ce jour, la version beta de BANCCO contient les données issues de 17 000 patients et plus de 35 000 CNV et sera rapidement enrichie. La collecte de données hétérogènes à partir de bases de données externes, d’outils de prédication et d’annotations d’experts permettra en outre de proposer à la communauté scientifique des données de qualité diagnostique enrichies de nombreuses annotations et d’une classification.